PCE │福建农林大学缪颖教授团队创建了深度神经网络TFBind模型可用于筛选转录因子-靶基因模块
2025-04-14 16:49:30
近年来各国科学家通过转录组测序(RNA-seq),染色质免疫共沉淀测序(ChIP-seq)和开放染色体组测序(ATAC-seq)积累了大量数据,建立多个数据库。但是,对于这些数据利用效率低,实验室耗费大量时间和经费来筛选目标基因。随着人工智能时代的到来,近年陆续报道了一些用于解决转录因子与目标基因之间相互作用的传统机器学习,如随机森林算法(张等人,2023年),仅涉及识别已知转录因子与目标DNA之间的相互作用。最新有文献报道,深度神经网络可用于识别转录因子结合位点(Ding 等人,2023年),例如,利用转录因子蛋白质构象与结合元件DNA 构象之间的亲和力数据构建了深度神经网络(Beak 等人,2024 年)。但是,蛋白结构信息往往数据量大、消耗内存,使得模型使用起来极为不便。
近日,福建农林大学缪颖课题组在植物学经典权威期刊Plant Cell & Environment上发表了题为“Deep neural network-mining of rice drought-responsive TF-TAG modules by a combinatorial analysis of ATAC-seq and RNA-seq”的研究论文,本研究在创建深度神经网络TFBind模型的基础上,利用该模型成功筛选了水稻干旱响应转录因子-靶基因模块。

该模型的链接URL:http://pypi.org/project/TFBind/
瞄准干旱胁迫对水稻产量的影响涉及全球粮食安全的问题,本研究进一步从公共数据库中获取了与非生物胁迫相关的水稻RNA-seq和ATAC-seq数据,利用WGCNA和ATAC-seq的综合分析,有效地鉴定出了与干旱时开放染色质区域相关的转录因子。通过TFBind模型测试,只有81%的转录因子与开放基因直接结合。通过这种方法,我们发现了15个与目标染色质开放区域相对应的干旱响应转录因子,这些转录因子主要富集在与蛋白质运输、蛋白质分配、氮化合物运输相关的条目中。这为干旱响应基因转录调控机制的研究提供基础。因此,这个模型为预测生物过程中的TF-TAG开放模块提供了一种有价值的工具。
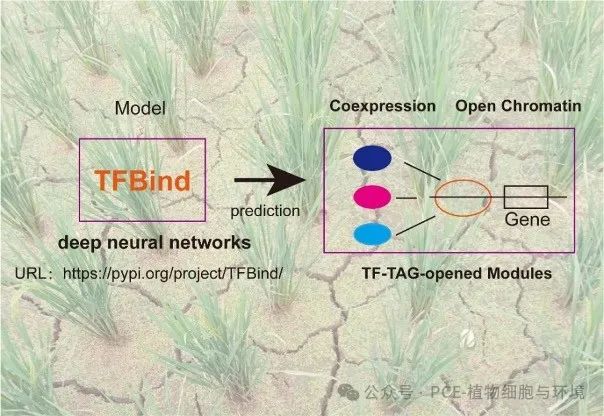
摘要图:通过深度神经网络模型TFBind结合WGCNA和ATAC-seq的数据综合分析挖掘水稻干旱响应转录因子-靶基因模块
该研究由福建农林大学缪颖教授团队完成,已毕业的硕士研究生刘经鹏和在读博士研究生石曦苗是论文的共同第一作者,缪颖教授和伍炳华教授为共同通讯作者。本研究得到了福建省自然科学基金重点项目(2021J02025)的资助。
福建农林大学缪颖教授团队围绕植物衰老的生物学基础,在分子细胞水平上解析植物细胞衰老的基因转录、表观修饰和细胞器间信号传导的机制,并进一步解析农作物水稻叶片和园艺作物花卉器官衰老和抗逆的生物学基础,其成果已多次发表于植物学知名期刊,包括Plant Cell,Molecular Plant,Journal of Integrative Plant Biology,Plant Physiology,Plant Journal等,为农作物高产优质育种和品质改良提供理论基础和技术支撑。
作者介绍:

刘经鹏(共同第一作者)
刘经鹏,目前在上海睿璟公司生物信息部门工作,本科软件工程专业,硕士生物工程专业。在本研究中完成神经网络模型TFBind的创建和大数据分析。

石曦苗,福建农林大学生命科学学院博士研究生,研究生期间主要从事“基于叶片衰老的水稻籽粒灌浆和干旱响应的分子机理”的研究。在本研究中完成水稻干旱响应TF-TAG-opened模块的EMSA验证。

缪 颖(通讯作者)


该期刊专注于发表最高质量的相关理论或实验文章和原创研究,因其能在植物科学领域中提供研究者们全新见解的特点而备受推崇。期刊由英国著名植物生理学家Harry Smith教授于1978年创立,由北京林业大学林金星教授和英国伯明翰大学 Christine Foyer教授担任主编。
期刊主页:wileyonlinelibrary.com/journal/pce
欢迎植物研究领域同仁投稿与转载,详情请通过微信或邮件联系本公众号维护团队。
撰稿 | 刘经鹏 曦苗
排版 | 张曦
审核 | 缪颖

